「倾向性评分匹配」(propensity score matching,PSM)是一种用来评估处置效应的统计方法。广义说来,它将样本根据其特性分类,而不同类样本间的差异就可以看作处置效应的无偏估计。
PSM主要是在随机对照试验(Randomized controlled trials,RCT)中用于衡量treat组和control组样本的其他各项特征(如年龄、体重、身高、人种等)的整体均衡性的度量。比如说研究一种药物对疾病的影响,在临床实验中,treat组和control组除了使用药物(安慰剂)不同外,其他的临床特征(如年龄、体重等)都应该基本是相似的,这样treat和control组才有可比性,进而才能验证药物的有效性。
一个例子
简单的看一个中文期刊的例子吧。
❝[1]侯松林,谢兴江,彭强,李利发,周何,周彤.结直肠癌肝转移病人原发灶切除的临床意义及其预后影响因素分析——基于SEER数据库的倾向得分匹配分析[J].临床外科杂志,2021,29(06):543-548.
❞

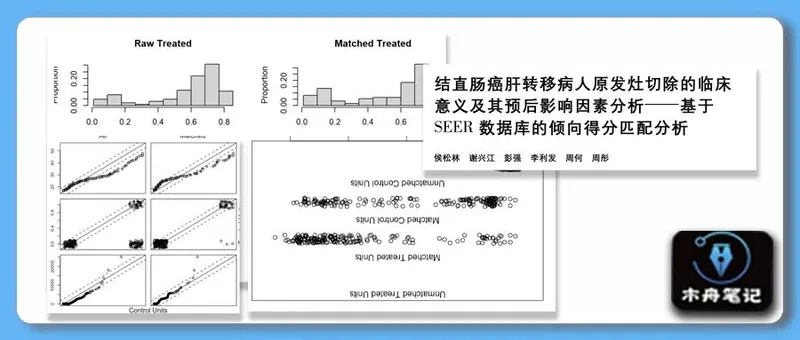
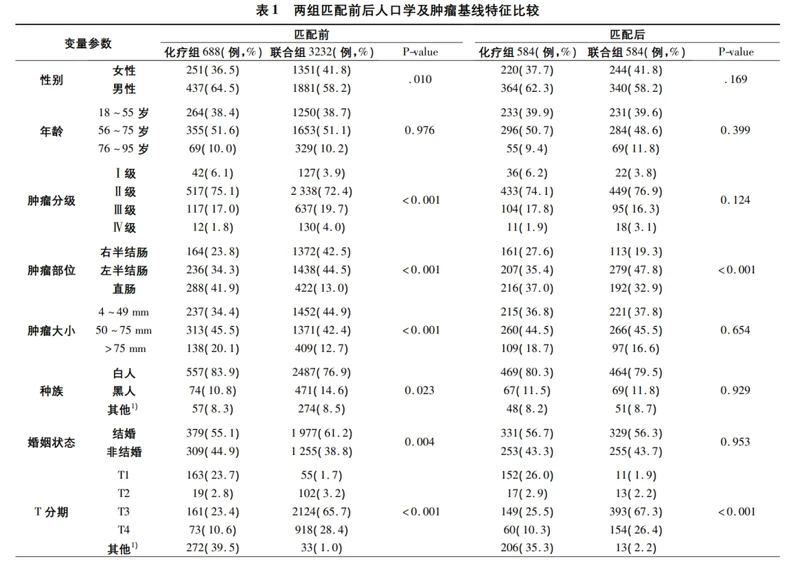
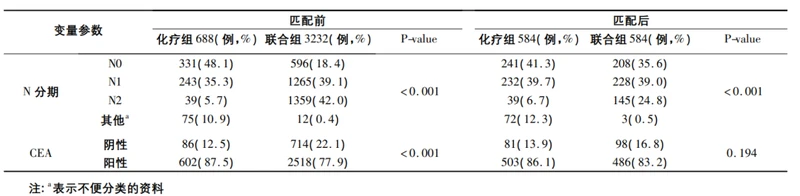
❝在纳入研究的两组病人中,病人性别、肿瘤分级、肿瘤部位、肿瘤大 小、种族、婚姻状态、T 分期、N 分期、CEA 等比较,差异有统计学意义( P < 0. 05) 。对匹 配后两组病例特征进行分析后显示,两组病例的倾向性评分分布具有一致性,并且匹配评分效果稳定可靠, 这说明匹配后两组的病例数据具有更好的可比性。基于此基础,比较匹配后的两组病人基线特征差异情况, 结果显示,两组病人基线特征差异明显缩小。
❞
PSM实战
# 安装和加载R包
install.packages("MatchIt")
library(MatchIt)
# 载入内置示例数据
data("lalonde")
head(lalonde)> head(lalonde)
treat age educ race married nodegree re74 re75 re78
NSW1 1 37 11 black 1 1 0 0 9930.0460
NSW2 1 22 9 hispan 0 1 0 0 3595.8940
NSW3 1 30 12 black 0 0 0 0 24909.4500
NSW4 1 27 11 black 0 1 0 0 7506.1460
NSW5 1 33 8 black 0 1 0 0 289.7899
NSW6 1 22 9 black 0 1 0 0 4056.4940❝❞研究假说是接受过职业培训会增加1978年的收入水平。 1. age:年龄,以岁为单位。 2. educ:学习时间,之前一共上过多少年学。 3. black:是否为黑人。 4. hispan:是否为西班牙裔。 5. married:是否已婚。 6. nodegree:是否没有毕业文凭。 7. re74:1974年的收入。 8. re75:1975年的收入。 9. re78:1978年的收入。 10. treat:1978年前是否接受职业培训。
先看一下未匹配前的基线数据
#install.packages("table1")
library(table1)
pvalue <- function(x, ...) {
# Construct vectors of data y, and groups (strata) g
y <- unlist(x)
g <- factor(rep(1:length(x), times=sapply(x, length)))
if (is.numeric(y)) {
# For numeric variables, perform a standard 2-sample t-test
p <- t.test(y ~ g)$p.value
} else {
# For categorical variables, perform a chi-squared test of independence
p <- chisq.test(table(y, g))$p.value
}
# Format the p-value, using an HTML entity for the less-than sign.
# The initial empty string places the output on the line below the variable label.
c("", sub("<", "<", format.pval(p, digits=3, eps=0.001)))
}
table1(~ age + educ + race + married + nodegree + re74 + re75 + re78| treat,
data=lalonde, overall=F, extra.col=list(`P-value`=pvalue))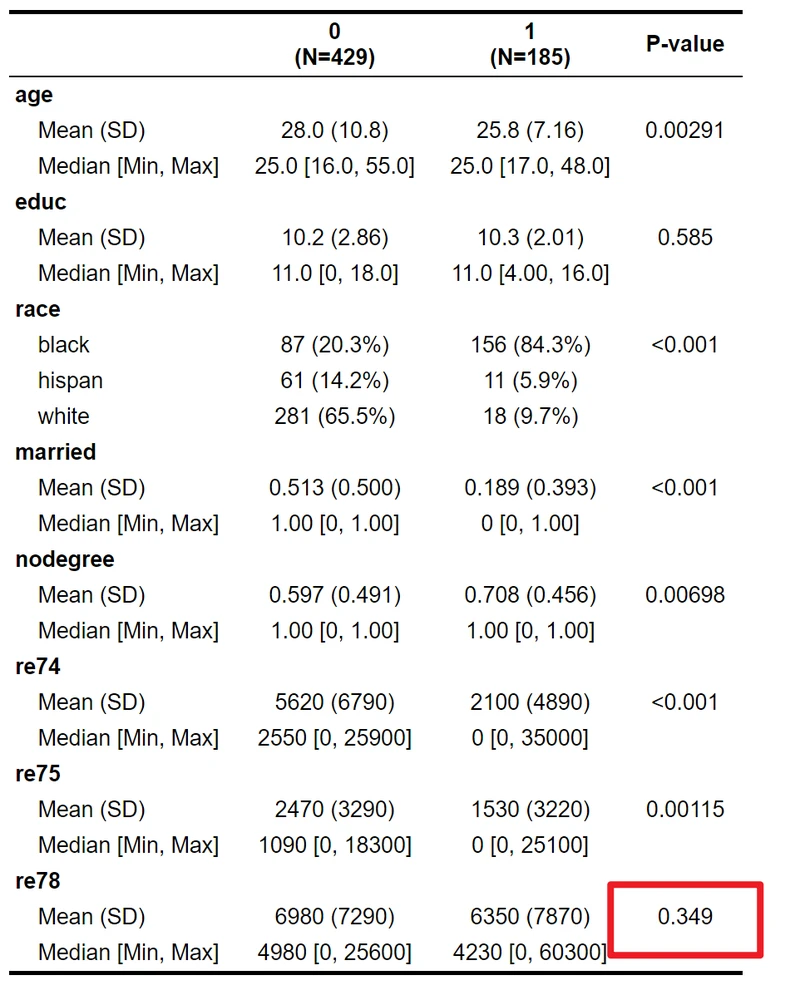
❝「可以看到组间并没有1978年收入没有差异,但是其他混杂因素均有较显著差异,现在就用R来行倾向性分析,匹配后可得到剔除混杂因素影响的样本。」
❞
匹配后基线数据
set.seed(50)
#PSM
m.out <- matchit(data = lalonde,
#以treat分组,匹配混杂因素(age,educ,black,hispan,married,nodegree,re74,re75)无差异的样本
formula = treat ~ age + educ + race + married + nodegree + re74 + re75
,
method = "nearest",
distance = "logit",
replace = FALSE,
caliper = 0.05)
#匹配后样本数据
lalonde_matched<-match.data(m.out)
table1(~ age + educ + race + married + nodegree + re74 + re75 + re78| treat,
data=lalonde_matched, overall=F, extra.col=list(`P-value`=pvalue))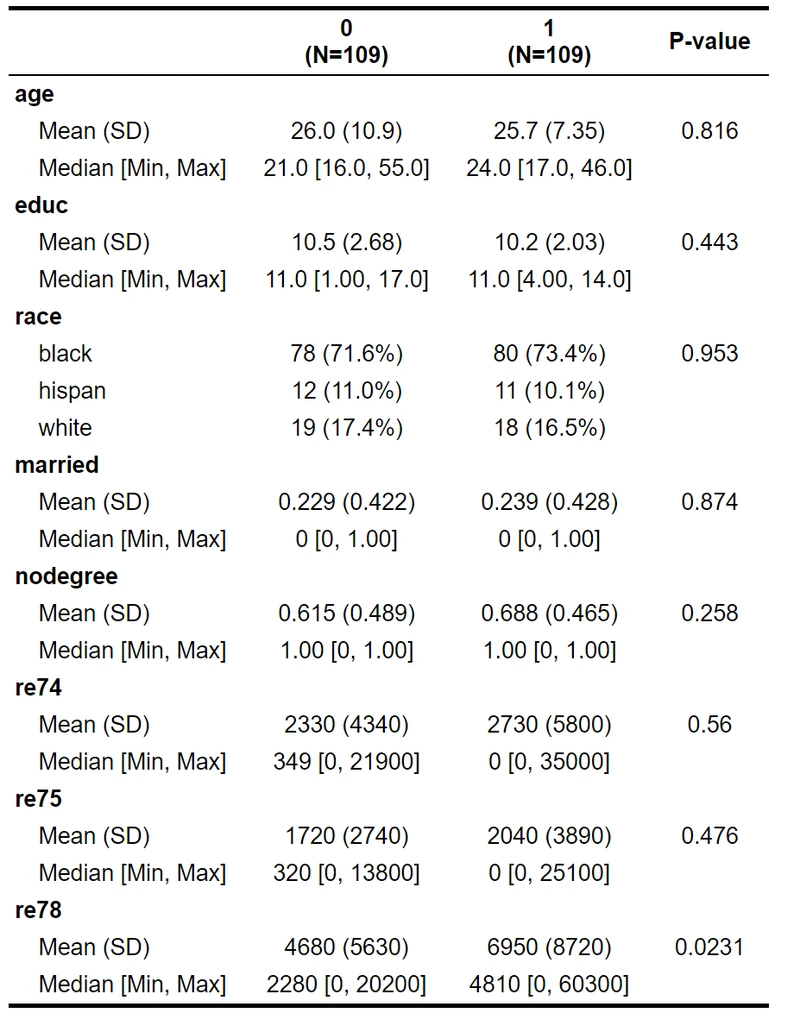
❝「经过倾向性评分匹配获得的数据组间其他混杂因素均无统计学差异,而职业培训组1978年收入较对照组明显升高,提示接受职业培训可以增加收入水平。」
❞
其实倾向性评分就是给通过结局变量和混杂因素「formula = treat ~ age + educ + race + married + nodegree + re74 + re75」做了一个回归分析,得到了distance就是该数据的倾向指数,然后依据倾向指数来均衡组间非试验因素的分布。
画出样本的倾向评分的分布情况
plot(m.out, type = "jitter", interactive = FALSE)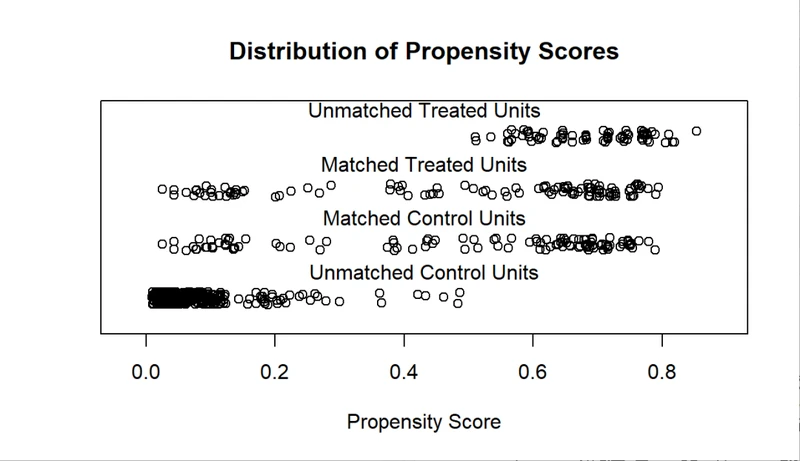
# 每个变量的分布
plot(m.out1, type = "qq", interactive = FALSE,
which.xs = c("age", "married", "re75"))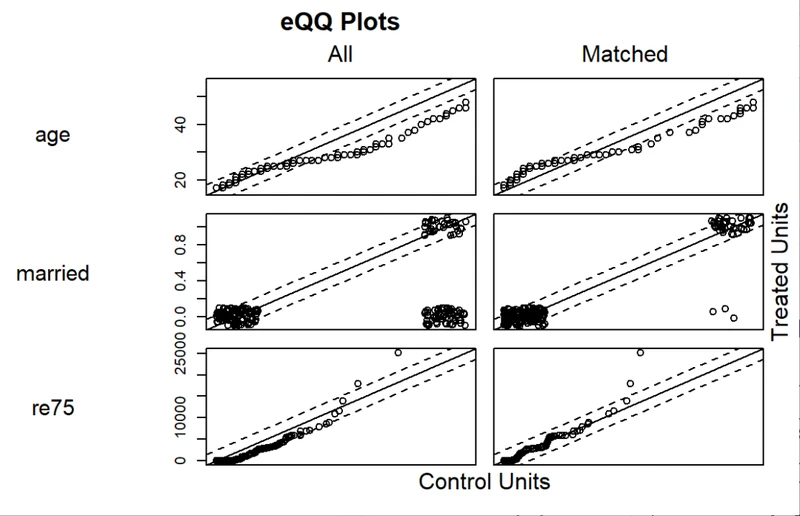
plot(m.out, type = "hist")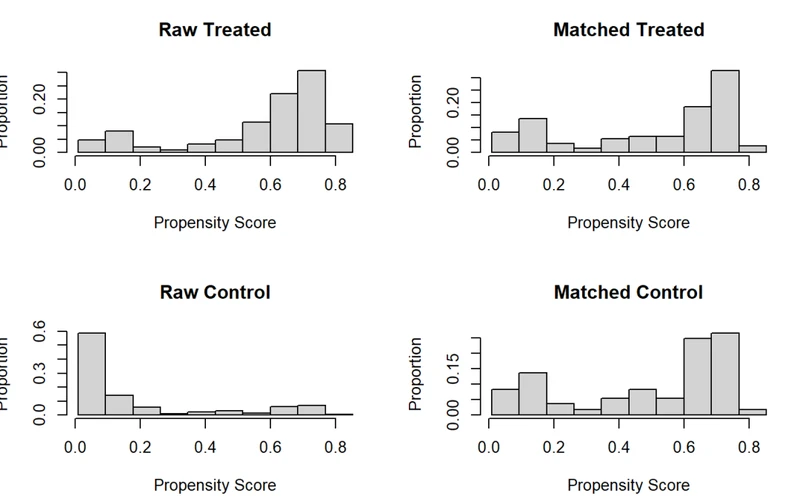
plot(summary(m.out))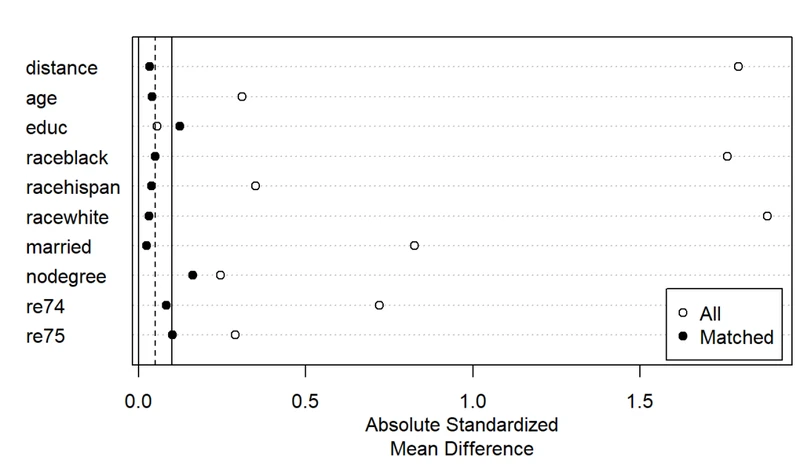
示例数据和代码领取
点赞、在看 本文,分享至朋友圈集赞25个并保留30分钟,截图发至微信mzbj0002领取。
「木舟笔记2022年度VIP可免费领取」。
木舟笔记2022年度VIP企划
「权益:」
「2022」年度木舟笔记所有推文示例数据及代码(「在VIP群里实时更新」)。
资源合集 木舟笔记「科研交流群」。
「半价」购买
跟着Cell学作图系列合集(免费教程+代码领取)|跟着Cell学作图系列合集。
「收费:」
「99¥/人」。可添加微信:mzbj0002 转账,或直接在文末打赏。

参考
MatchIt: Getting Started (r-project.org)(https://cran.r-project.org/web/packages/MatchIt/vignettes/MatchIt.html)
往期内容
CNS图表复现|生信分析|R绘图 资源分享&讨论群!
组学生信| Front Immunol |基于血清蛋白质组早期诊断标志筛选的简单套路
(免费教程+代码领取)|跟着Cell学作图系列合集
Q&A | 如何在论文中画出漂亮的插图?
跟着 Cell 学作图 | 桑葚图(ggalluvial)
R实战 | Lasso回归模型建立及变量筛选
跟着 NC 学作图 | 互作网络图进阶(蛋白+富集通路)(Cytoscape)
R实战 | 给聚类加个圈圈(ggunchull)
R实战 | NGS数据时间序列分析(maSigPro)
跟着 Cell 学作图 | 韦恩图(ggVennDiagram)

