一、文章背景介绍
DiffHopp模型发表在ICML 2023 Workshop on Computational Biology(简称:2023 ICML-WCB)上的文章。第一作者是剑桥计算机系的Jos Torge。

DiffHopp是一个专门针对骨架跃迁任务而训练的E3等变条件扩散模型。此外,DiffHopp使用了更具有几何表达力的图神经网络GVP模型。DiffHopp模型针对给定蛋白质-配体复合物,使用等变扩散模型从以官能团和蛋白质袋为条件的骨架分布中对骨架进行采样。 所得骨架与官能团合并以形成骨架跃迁配体。
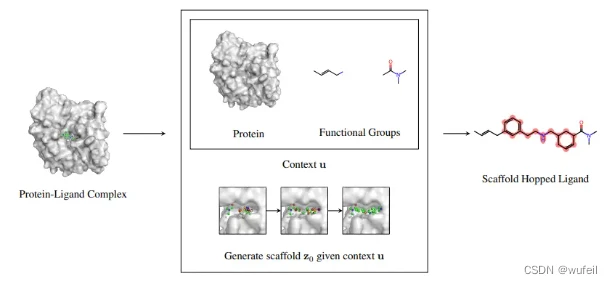
(有趣的是,我在生成分子的代码中,并没有看到输入官能团的参数。官能团不知道是怎么识别的?估计在数据处理的代码中,需要仔细看看)
关于骨架跃迁任务,作者认为是:保留了原始支架的基本分子特征,同时引入新的化学元素或结构特征以增强效力、选择性或生物利用度。分子结构中间区域都会发生改变,但是做两侧的基团不发生变化或者少量变化。换句话说,骨架跃迁需要通常需要重新设计大部分分子。如下图所示。

这一点与我们之前介绍的其他模型明显不同,Difflinker,Dlinker,Delinker, DEVELOP都是仅修改指定的分子中间一小段。而DiffHopp是不能指定特定的分子区域,同时分子修改区域很大,两侧的功能基团的位置也会发生变化。但这一点其实是不利的,因为在做骨架跃迁项目时,我们希望生成的分子具有晶体分子一样的性质,里面就包括,类似的结合模式和形状。当功能基团的位置发生变化了,很可能原先的相互作用模式被改变了,生成的分子也就没有被相信的理由了。没办法,项目就是这样子,可以相信一个可能对的东西,但是绝对不会信一个AI的观察,毕竟现在AI的观察还是不完善的。
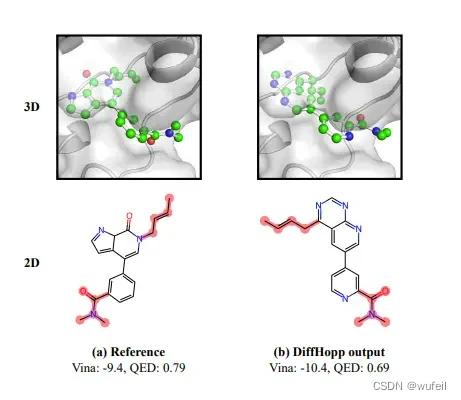
作者以6bqd作为例子,生成了10个分子,然后进行VINA打分。最优打分的分子的docking score为-10.4明显小于参考分子的-9.4,如下图所示。功能基团为红色标记区域。
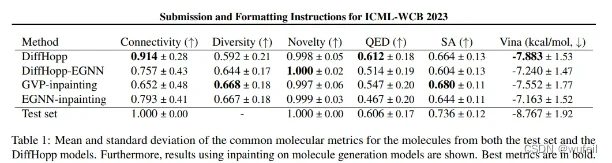
作者对测试集体系都做了DiffHopp模型的骨架跃迁设计,并使用vina评估打分,结果如下表。DiffHopp模型的结果要优于GVP和ENGG等模型使用inpainting模式的结果,特别是在分子成药性QED和结合力 vina score上。同时也验证了使用GVP模型的扩散模型 (DiffHopp)比使用ENGG模型的扩散模型(DiffHopp-EGNN)生成的分子更具有优势(DiffHopp vs. DiffHopp-EGNN)
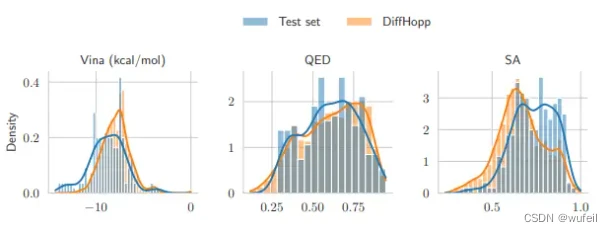
但是根据作者的结果,生成分子的QED、SA分数,和vina score打分上仅比测试集的数据分布好一些,说明生成的骨架跃迁分子要优于参考分子,模型在某些体系上捕捉到了额外的蛋白分子相互作用。但结果并不显著。
二、代码复制与环境安装
复制项目代码
git clone https://github.com/jostorge/diffusion-hopping.git创建conda环境和安装相应的包
#安装环境
conda env create -f environment.yml
#激活环境
conda activate diffusion_hopping注意,可能由于cuda版本以及系统的原因,导致安装的pytorch可能是cpu版本需要重新安装pytorch。
安装reduce
reduce是用于蛋白质中H原子处理的包,可以加氢也可以删除氢,同时也可以对氨基酸做一些处理,例如HIS。reduce的安装可以参考:GitHub - rlabduke/reduce: Reduce - tool for adding and correcting hydrogens in PDB files.
也可以安装AMBERTools,使用AMBERTools中的reduce替代, 然后将reduce软连接出来。
AMBERTools的安装方法:
conda install -c conda-forge ambertools=23软连接方法:在~/.bashrc文件中添加如下行
alias reduce='~/anaconda3/envs/AmberTools23/bin/reduce'安装Qvina,选装。(仅在分子评估部分使用)
这里略过
安装mgltools
conda install mgltools -c bioconda三、案例测试(1)
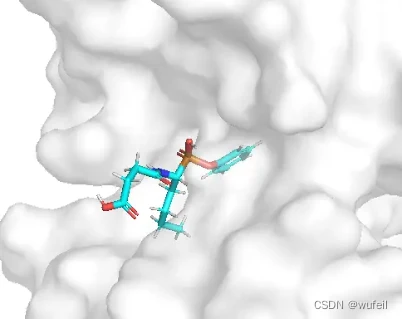
作者在代码中自带了一个体系,1a0q,路径为:
./tests_data/complexes/1a0q口袋和分子结构见下图:
使用如下命令生成100个新骨架跃迁分子:
python generate_scaffolds.py \
--input_molecule ./tests_data/complexes/1a0q/ligand.sdf \
--input_protein ./tests_data/complexes/1a0q/protein.pdb \
--num_samples 100 \
--output test_data_results生成的分子保存在自动创建的 ./test_data_results文件夹内。其中,只产生了100个sdf文件,对应100个分子。生成过程不存在不合格的分子。
先简单查看几个生成的分子,如下图。生成的分子与参考分子并没有明显的变化,只有在口袋内侧的苯环发生了角度旋转。这一结果来说,DiffHopp模型对于这个体系的骨架跃迁就失败了。很有可能是代码在识别scaffold区域时,只识别到了苯环,而在生成scaffold时也只生成了苯环。
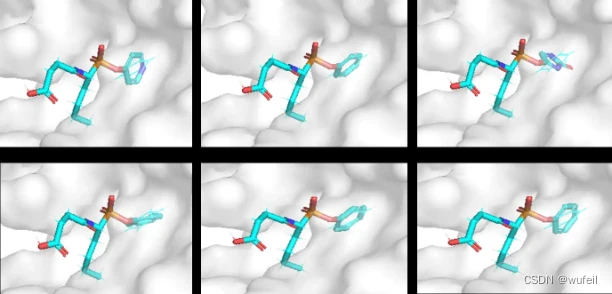
另外,所有生成的分子均为23个重原子数,与参考分子完全相同。侧面也说明,DiffHopp生成的分子和SBDD等分子生成模型,或者Difflinker等骨架跃迁模型的inpainting模式类似,并不改变生成分子的原子数量。模型时调整原子的位置及其元素种类,而生成新的分子。
生成分子的视频如下:
DiffHopp_1a0q
